Ómicas
Dos marcadores moleculares asociados a la acumulación de sacarosa fueron utilizados en la validación de técnicas de genotipado. Las técnicas de secuenciación dirigida resultaron promisorias para identificar marcadores en el genoma poliploide de la caña de azúcar. Avance de investigación.
Autores: Fernando S. Aguilar1, Alejandra Londoño1, 2, Carolina Saavedra2, Hugo Arley Jaimes1, Jhon Henry Trujillo1, Jershon López Gerena1 y John Jaime Riascos1

La alianza ÓMICAS, Optimización Multiescala In-silico de Cultivos Agrícolas Sostenibles, es un programa de cooperación en el foco estratégico de Alimentos en Colombia, diseñado para brindar soluciones agrícolas en materia de seguridad alimentaria y sostenibilidad productiva mediante la investigación y la educación en ciencias ómicas.
En cinco años de actividades (2017-2022), entre los resultados se cuenta con la validación de técnicas analíticas de genotipado, dirigidas a la secuenciación del genoma completo (WGS) y capaces de reproducir marcadores SNP, es decir, polimorfismos de un solo nucleótido. La experimentación genómica se llevó a cabo después de secuenciar 220 genotipos de caña de azúcar (Saccharum spp.) del banco de germoplasma de Cenicaña, y alinear la información al genoma completo de la variedad CC 01-1940. Con esta información, se realizaron asociaciones y se seleccionaron SNP relacionados con la acumulación de sacarosa.
¿Qué se espera de las técnicas de genotipado?
- Identificar con precisión la posición de un marcador tipo SNP en el genoma poliploide de la caña de azúcar.
- Estimar la dosis alélica de marcadores SNP en cada posición del genoma, es decir, el número de alelos de referencia (aquellos que son iguales a los del genoma de referencia CC 01-1940) y alelos alternativos (diferentes a los de CC 01-1940).
- Aumentar la capacidad para procesar un gran número de genotipos (muestras de ADN) con rapidez y confiabilidad.
- Reducir los costos de genotipado y ganar eficiencia en los estudios de genómica.
Asistir el mejoramiento vegetal en la producción de genotipos de alta productividad.
Probando técnicas de amplificación y secuenciación
Cenicaña evaluó tres técnicas de genotipado, dos de ellas basadas en amplificación del ADN (KASP y ddPCR) y una tercera basada en secuenciación (Flex-seq).
- KASP (Kompetitive Allele Specific PCR) se fundamenta en una PCR competitiva alelo-específica que permite diferenciar variaciones en un solo nucleótido por cambios en los niveles de fluorescencia, de modo que es posible asignar la clase genotípica a cada individuo (i.e., homocigoto o heterocigoto) según el conteo de los alelos de referencia y los alternativos.
- La ddPCR (Droplet Digital PCR) incluye la partición del ADN en múltiples repeticiones mediante la emulsión de agua y aceite para la formación de microgotas (más de 18,000, cada una de volumen cercano a 1 nanolitro). Estas microgotas se analizan por PCR digital para incrementar los niveles de detección de marcadores SNP al usar sondas fluorescentes específicas.
- Flex-seq® es una metodología basada en secuenciación dirigida a las regiones adyacentes del marcador SNP de interés (aproximadamente 400 nucleótidos). Esta metodología, desarrollada por la compañía Rapid-Genomics, tiene la ventaja de detectar la secuencia de interés con mayor precisión y agilidad.
Las tres técnicas permitieron estimar la dosis alélica de SNP en cada una de las posiciones del genoma de los individuos evaluados.
Validando la reproducibilidad de las dosis alélicas
Las dosis alélicas en caña de azúcar pueden variar entre 0 y 10 debido a la poliploidía de la especie, un rango muy superior al de organismos diploides como el maíz o el arroz que varían entre 0 y 2.
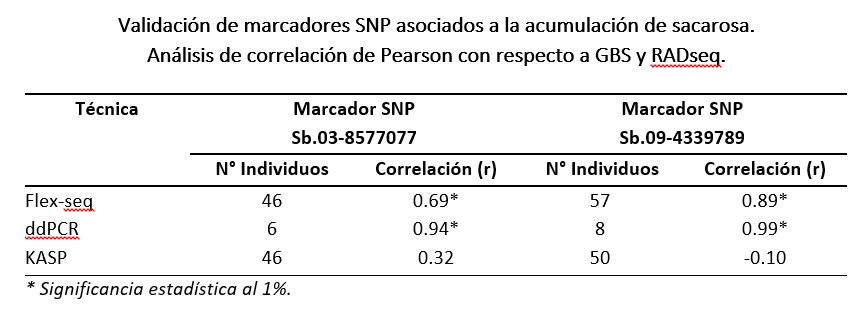
Así, en aras de verificar el grado de reproducibilidad de marcadores SNP con las nuevas técnicas, se llevó a cabo el experimento de validación para comparar las dosis alélicas obtenidas con cada una (KASP, ddPCR y Flex-seq) y las generadas en años anteriores por medio de GBS (genotipado por secuenciación) y RADSeq (secuenciación de ADN asociada al sitio de restricción). Para el efecto se estimó el coeficiente de correlación de Pearson, una medida de dependencia lineal entre dos variables aleatorias cuantitativas que no resulta afectada por la escala de medida de las variables.
Conclusiones
- Flex-seq y ddPCR presentaron coeficiente de correlación (r) mayores que 0.69, lo que sugiere que ambas técnicas permiten reproducir los resultados obtenidos mediante análisis GBS y RADseq.
- Para KASP, se observó una correlación baja con respecto a GBS y RADseq, lo que indica baja reproducibilidad de la técnica para secuencias SNP. En este caso, ninguno de los marcadores tuvo una fluorescencia distintiva de un organismo decaploide, por lo que se utilizó una asignación típica de organismos diploides. Sin embargo, este supuesto de diploidía dio como resultado un coeficiente de correlación menor a 0.32, indicativo de baja reproducibilidad de la técnica.
- Flex-seq tiene un menor costo en el procesamiento de los datos y presenta alta capacidad de identificar con precisión la posición de un marcador SNP en el genoma poliploide de la caña de azúcar, lo que hace que esta técnica sea idónea para la validación de marcadores moleculares de importancia económica en poblaciones de caña de azúcar.
Finalmente, acorde con los resultados, se decidió evaluar alternativas de secuenciación dirigida, es decir técnicas del tipo Flex-seq, buscando reducir los costos de genotipado.
| SNP | Single Nucleotide Polymorphis | Polimorfismo de un solo nucleótido | |
| WGS | Whole Genome Sequencing | Secuenciación de genoma completo | (Neves et al., 2012) Neves, H. H. R., Carvalheiro, R., & Queiroz, S. A. (2012). A comparison of statistical methods for genomic selection in a mice population. BMC Genetics, 13. https://doi.org/10.1186/1471-2156-13-100 |
| GBS | Genotyping-By-Sequencing | Genotipado por secuenciación | (Elshire et al., 2011) Elshire, R. J., Glaubitz, J. C., Sun, Q., Poland, J. A., Kawamoto, K., Buckler, E. S., & Mitchell, S. E. (2011). A robust, simple genotyping-by-sequencing (GBS) approach for high diversity species. PLoS ONE, 6(5), 1–10. https://doi.org/10.1371/journal.pone.0019379 |
| RADSeq | Restriction-site Associated DNA Sequencing | Secuenciación de ADN asociada al sitio de restricción | (Davey et al., 2011) Davey, J. W., Hohenlohe, P. A., Etter, P. D., Boone, J. Q., Catchen, J. M., & Blaxter, M. L. (2011). Genome-wide genetic marker discovery and genotyping using next-generation sequencing. Nature Reviews Genetics, 12(7), 499–510. https://doi.org/10.1038/nrg3012 |
| KASP | Kompetitive Allele Specific PCR | PCR competitiva alelo-específica | (He et al., 2014) He, C., Holme, J., & Anthony, J. (2014). SNP genotyping: the KASP assay. Methods in molecular biology (Clifton, N.J.), 1145, 75–86. https://doi.org/10.1007/978-1-4939-0446-4_7 |
| ddPCR | Droplet digital PCR | PCR digital de gotas | Hindson et al., 2011 |
| Flex-seq | secuenciación dirigida a las regiones adyacentes del SNP de interés |
____________________
1 Centro de Investigación de la Caña de Azúcar, Cenicaña
2 Pontificia Universidad Javeriana, Cali










